每当拿到一个质粒图谱,第一反应就是一脸懵,这是什么玩意?这些ori、RBS、tet是什么鬼?这些箭头代表什么,转录的方向吗?为什么都不相同,不会“撞车”么?这玩意究竟怎么看啊?
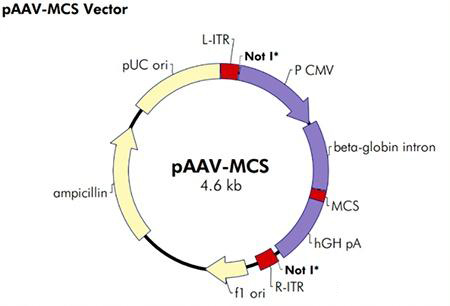
看懂质粒图谱的4个要点
质粒的主要构成元件
1 复制起始位点Ori
即控制复制起始的位点。Ori的箭头指复制方向,其他元件标注的箭头多指转录方向(正向)。
图谱上只有一个ori,表示质粒是原核克隆及表达质粒, 图谱上有两个ori,则表示该质粒是穿梭质粒,既可以在原核也可以在真核中复制。
图谱中的AmpR、KanaR等表示质粒载体中的筛选标签,多为抗生素抗性基因,方便后续通过抗生素筛选阳性克隆。特点是单词最后会以大写R或上标r结束。
只在一种宿主中复制的质粒仅具有一种抗性筛选标记,如原核克隆载体和原核表达载体;能够在两种类型宿主中复制和表达的载体会具有两种筛选标记,如穿梭质粒。
可以便于加以检测
Ampr:水解β-内酰胺环,解除氨苄的毒性。
tetr :可以阻止四环素进入细胞。
camr:生成氯霉素羟乙酰基衍生物,使之失去毒性。
neor(kanr):氨基糖苷磷酸转移酶,使G418(卡那霉素衍生物)失活。
hygr:使潮霉素β失活。
3 多克隆位点
图谱中的MCS区或者许多内切酶集中的部分,就是质粒的多克隆位点。多克隆位点为一系列限制性内切酶酶切位点,是外源DNA的插入位点,一般可通过酶切后连接的方式将外源DNA插入质粒。
图谱中的MCS区或者许多内切酶集中的部分,就是质粒的多克隆位点。多克隆位点为一系列限制性内切酶酶切位点,是外源DNA的插入位点,一般可通过酶切后连接的方式将外源DNA插入质粒。多克隆位点一般位于转录启动和转录终止信号之间。
4 其他元件
质粒载体中除复制起始位点、筛选标记、多克隆位点外,还具有其他一些表达或调控元件,如转录调控元件、翻译调控元件和融合表达标签蛋白等。
P/E:启动子/增强子,Terms:终止信号;加poly(A)信号:可以起到稳定mRNA的作用等等。
解读质粒图谱
知道了这些基础知识,再回过头看看如何阅读质粒图谱:
1 看箭头方向
一般质粒都会有箭头,箭头有两种解释:一种是转录方向,转录方向主要是由启动子开始的一个箭头,是启动子启动序列的顺序。另一种是复制起始位点的方向,复制起始位点就是该质粒在大肠杆菌等细菌或真菌中DNA复制的一个方向。
2 看各种标签
① ori:DNA复制起始位点,一般在大肠杆菌中是pUC类的,还有一种F1启动子(图上的f1ori)代表的是噬菌体的复制起始方向,只能复制出单链的DNA,可以用来测序。

②P:一般是代表启动子,这个质粒上用的是一个CMV启动子,来启动后面的表达蛋白。常见的启动子有:CMV、EF1(启动长片段),H1、U6(启动短片段,比如shRNA)。

③抗性基因:最常见的是:Amp(氨苄青霉)、Tet(四环素)、Cmr(氯霉素)、SM(链霉素)、Hyg(潮霉素)。

④pA:转录终止位点。
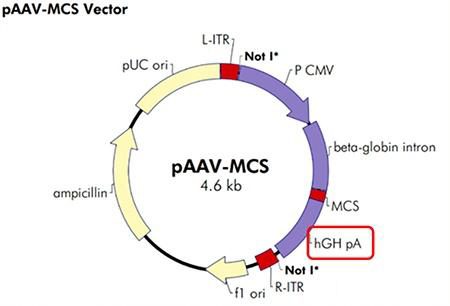
⑤ 报告基因:通常会有一两个蛋白,被用作报告基因。比如常见的copGFP(绿色荧光蛋白),Puro(嘌呤霉素),Lacz(乳糖操纵子)等等。和抗性基因不同,报告基因是为了显示过表达或是敲减的基因是否正常运作。有的报告基因会融合在蛋白中表达,有的会另外用一个独立的启动子进表达(比如shRNA的质粒中)。
3 看MCS(多克隆位点)
多克隆位点为一系列限制性内切酶酶切位点,是外源DNA的插入位点,一般可通过酶切/连接的方式将外源DNA插入质粒。

4 看外源DNA片段
质粒一般只能容纳小于10Kb的外源DNA片段。一般来说,外源DNA片段越长,越难插入,越不稳定,转化效率越低。
5 是否含有表达系统元件
即启动子-核糖体结合位点-克隆位点-转录终止信号。这是用来区别克隆载体与表达载体。克隆载体中加入一些与表达调控有关的元件即成为表达载体。选用那种载体,还是要以实验目的为准绳。
实验外包 想了解更多请关注:http://www.do-gene.cn

